題目:Pervasive Chromatin-RNA Binding Protein Interactions Enable
RNA-Based Regulation of Transcription
期刊:Cell
產品:FIP1L1 antibody (A5016)
物種:人
應用:ChIP-Seq
一、研究背景
RNA結合蛋白(RNA Binding Protein,RBP)在轉錄後的剪接加工、修飾、轉運、細胞定位、穩定性、翻譯和降解等RNA代謝過程中發揮著重要的調控功能,但近來對RNA可交聯蛋白質的觀察揭示了大量的經典和非經典RBP。目前已知一些典型的DNA結合蛋白能夠結合RNA,現已擴展到許多轉錄因子(Transcription Factors , TFs),例如CTCF; DNA損傷修復酶,例如XRCC5 / Ku80,以及轉錄複合物,例如PRC2。保守估計人類基因組目前編碼的RBPs多達1500種,鑑於哺乳動物細胞中存在如此巨大的RBP庫,現在需要研究和探索它們新的功能。而且越來越多的證據表明,轉錄調控和染色質活性在很大程度上涉及調節類的RNA,儘管多個RBPs與轉錄調控有關,但其在轉錄水平的調控功能和機制尚不清楚。
武漢大學肖銳研究員與美國加州大學聖地亞哥分校(UCSD)付向東教授課題組研究成果榮登國際頂級期刊Cell雜誌,該研究刷新了我們對RNA結合蛋白的傳統認知,揭示RNA結合蛋白是一類新的轉錄因子或輔助因子的新概念,可通過RNA依賴的染色質相互作用直接參與轉錄調控,開闢了RNA結合蛋白功能研究的新領域。肖銳研究員為共同第一作者和通訊作者,加州大學聖地亞哥分校陳加餘博士和梁徵宇博士為共同第一作者,付向東教授為共同通訊作者。該論文的共同作者還包括武漢大學周宇教授和羅大極副教授、清華大學魯志教授、張奇偉教授和陳陽助理研究員、廈門大學劉文教授、美國MIT Christopher Burge教授、UCSD Gene Yeo教授和康涅狄格大學Brenton Graveley教授和加拿大蒙特利爾大學Eric Lecuyer教授。
二、研究結果與內容
針對特定RBP參與轉錄和共轉錄RNA加工的這一新興領域,付向東等團隊參加了ENCODE項目,分別在HepG2和K562細胞上挑選了有特異性抗體且有核定位的58和45個RBP,並通過ChIP-seq(染色質免疫沉澱測序)技術大規模檢測,肖銳研究員等意外地發現約60%的RNA結合蛋白與染色質有廣泛的相互作用,並且放大在基因啟動子和增強子區域。
Figure 1.General Features of
Chromatin-Associated RBPs.
該研究目前只檢測了人類基因組中不到5%的RNA結合蛋白(目前RBPs大概有1500種),卻發現約40%的活性染色質區域和約80%的活性啟動子區域都存在RNA結合蛋白的特意性結合,這提示RNA結合蛋白在染色質水平存在著廣泛的功能和作用。
Figure 2.Distinct RBP-Chromatin Interaction
Patterns on Different Promoter Classes.
作者在HepG2細胞中進行RBP基因剔除,比較基因表達在穩態前後的變化,通過GRO-seq(Global Nuclear Run-On測序)技術和RNA-seq技術發現野生型細胞系大多數RBP與啟動子的相互作用與靶基因轉錄活性相關,基因剔除後,幾乎所有的RBP都在穩定狀態下影響基因表達。為了進一步判斷誘導基因表達是否與他們的啟動子相關,作者通過GRO-seq證實至少有6個RBP(RBM22, XRCC5, RBM25, HNRNPK, HNRNPLL和 U2AF1)直接參與了轉錄調控過程。
Figure 3.Correlation between RBP-Promoter
Interaction and Gene Expression.
通過NMF(Negative Matrix Factorization)方法進一步分析獲得的高質量RBPs ChIP-seq數據與相同細胞系中可利用的TFs ChIP-seq數據,揭示了許多共同結合事件,從而為TFs和RBPs的協同作用提供了證據。此外,研究發現RBPs如同TFs一樣偏向於結合人類基因組中HOT(High-occupancy Target)區域。
Figure 4.Integrated Analysis of
Chromatin-Associated RBPs and TFs in HepG2 Cells
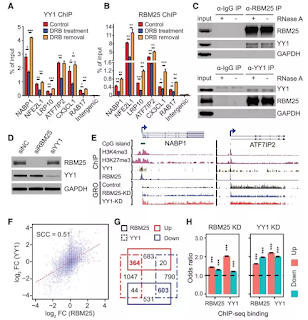
5、YY1和RBM25在轉錄中的功能協同作用該研究重點闡釋了RNA結合蛋白RBM25(參與剪接調節的RBP)通過調控轉錄因子YY1(已知的RNA依賴性TF)與染色質的結合,進而調控染色質結構和轉錄激活與抑制的分子機制, RBM25耗竭減弱了所有YY1依賴性活性,包括染色質結合,DNA環化和轉錄,全面展示了特異性的RNA結合蛋白介導的轉錄因子YY1的功能調控作用。
Figure 5.Co-regulation of Gene Expression
by YY1 and RBM25 in HepG2 Cells.
文章小結
該研究通過大規模RNA結合蛋白 ChIP-seq分析,證實了RNA結合蛋白廣泛存在於人類基因組中的活躍染色質區域。與轉錄因子一樣,RNA結合蛋白同樣顯示出對基因組中HOT區域的強烈偏好,特別是基因啟動子,其中它們的關聯通常與轉錄輸出相關。同時發現提出了RNA結合蛋白作為轉錄因子或輔助因子調控轉錄的新概念,開闢了RNA結合蛋白功能研究的新領域,刷新了我們對RNA結合蛋白的傳統認知。
三、RBP抗體案例展示
文獻參考
1. Akdemir, K.C., and Chin, L. (2015).
HiCPlotter integrates genomic data with interaction matrices. Genome Biol. 16,
198.
2. Aso, T., Vasavada, H.A., Kawaguchi, T.,
Germino, F.J., Ganguly, S., Kitajima, S., Weissman, S.M., and Yasukochi, Y.
(1992). Characterization of cDNA for the large subunit of the transcription
initiation factor TFIIF. Nature 355, 461–464.
3. Baltz, A.G., Munschauer, M.,
Schwanhausser, B., Vasile, A., Murakawa, Y., Schueler, M., Youngs, N.,
Penfold-Brown, D., Drew, K., Milek, M., et al. (2012). The mRNA-bound proteome
and its global occupancy profile on protein-coding transcripts. Mol. Cell 46,
674–690.
4. Bao, X., Guo, X., Yin, M., Tariq, M.,
Lai, Y., Kanwal, S., Zhou, J., Li, N., Lv, Y., Pulido-Quetglas, C., et al.
(2018). Capturing the interactome of newly transcribed RNA. Nat. Methods 15,
213–220.
5. Barutcu, A.R., Maass, P.G., Lewandowski,
J.P., Weiner, C.L., and Rinn, J.L. (2018). A TAD boundary is preserved upon
deletion of the CTCF-rich Firre locus. Nat. Commun. 9, 1444.
6. Benoit Bouvrette, L.P., Cody, N.A.L.,
Bergalet, J., Lefebvre, F.A., Diot, C., Wang, X., Blanchette, M., and Le ´
cuyer, E. (2018). CeFra-seq reveals broad asymmetric mRNA and noncoding RNA distribution
profiles in Drosophila and human cells. RNA 24, 98–113.
7. Bentley, D.L. (2014). Coupling mRNA
processing with transcription in time and space. Nat. Rev. Genet. 15, 163–175.
8. Bregman, A., Avraham-Kelbert, M.,
Barkai, O., Duek, L., Guterman, A., and Choder, M. (2011). Promoter elements
regulate cytoplasmic mRNA decay. Cell 147, 1473–1483.
9.
Cassiday, L.A., and Maher, L.J., 3rd. (2002). Having it both ways:
transcription factors that bind DNA and RNA. Nucleic Acids Res. 30, 4118–4126.














沒有留言:
張貼留言