導讀
真核生物細胞核內展開長度為2-3米的DNA如何折疊在直徑僅為數微米的細胞核中,並有序地調控基因組的功能?儘管經歷了多年的研究,但這目前依然是生命科學中尚未解決的重要科學問題之一。
2017年12月22日,復旦大學基礎醫學院和生物醫學研究院雙聘PI文波教授課題組在國際知名期刊Genome Research上發表題為“The nuclear matrix protein HNRNPU maintains 3D genome architecture
globally in mouse hepatocytes”的研究論文。該研究在核基質蛋白對染色質高級結構的全局性調控作用方面取得重要進展。文波教授為本文通訊作者,博士生範輝和呂品為第一作者。
本文中重要靶點H3K9me3抗體(貨號A2360)來自ABclonal公司,成功應用於WB和IF。
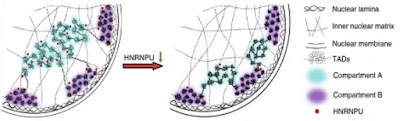
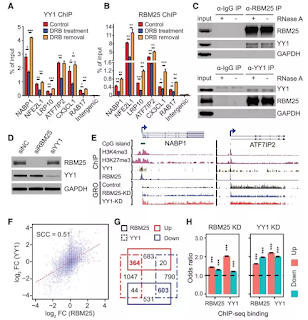
課題組研究人員用核型維持完整的小鼠肝臟細胞建立細胞模型,通過Hi-C (High-throughput/resolution chromosome conformation capture)、DamID (DNA adenine methyltransferase identification)、ChIP-Seq、RNA-Seq等高通量定序技術結合光學顯微鏡以及電鏡技術,系統研究了核基質蛋白HNRNPU (Heterogeneous Nuclear Ribonucleoprotein U)對3D genome(三維基因組結構)的調控功能。
實驗發現當HNRNPU的表達下調時,染色質和核纖層之間的相互作用,即核纖層關聯域(Lamina-associated
domains,LADs)發生了巨大的變化,致使16.6%的基因組由非LADs轉變為LADs區域,電鏡結果也表明染色質向核邊緣重分佈。
同時發現有7.5%的基因組發生了Compartment(間隔)的類型轉變,Compartment和TAD(Topologically Associated
Domains,拓撲結構域)內部及其之間的長距離相互作用也發生了顯著變化。此外,46%的TAD邊界強度及58%的染色質環強度也隨之降低。通過ChIP-seq實驗證明HNRNPU主要結合在活性染色質區域,而且80%的結合位置與CTCF或者RAD21相互重疊。
該項研究系統解析了HNRNPU對染色質高級結構的全局調控功能,並暗示核基質對3D genome的普遍性組織作用,為揭示高級結構染色質形成及維持的分子機制提供了新依據。
The working model of chromatin high structure
regulated by HNRNPU
ABclonal從成立之初,一直致力於表觀修飾型抗體研發,為科研工作者提供更多的表觀研究工具。
在哺乳動物基因組中,一個核體由H2A、H2B、H3、H4各兩分子組成的八聚體和200bp左右的DNA以及一個分子的組蛋白H1組成。游離在外的組蛋白N端會在相關酶作用下發生甲基化、乙酰化、磷酸化、腺苷酸化、泛素化、ADP核糖基化等多種修飾,這些修飾都會影響基因的轉錄活性。研究最熱門的是組蛋白的甲基化修飾,其次是乙酰化修飾。組蛋白甲基化是由組蛋白甲基轉移酶(Histonemethyl Transferase,HMT)催化完成,可發生在組蛋白的賴氨酸(K)和精氨酸(R)殘基上,賴氨酸殘基能夠發生單、雙、三甲基化,而精氨酸殘基能夠發生單甲基化、對稱和不對稱雙甲基化。組蛋白乙酰化則是由乙酰轉移酶介導,人類基因組編碼有六十多種組蛋白乙酰轉移酶,催化組蛋白的賴氨酸殘基上發生單乙酰化。
組織蛋白八聚體結構示意圖
組織蛋白常見修飾位點分佈
ABclonal組蛋白修飾型靶點抗體
ABclonal表觀修飾型抗體優勢
特異性高:抗體採用交叉純化,短肽純化,多肽矩陣檢測抗體特異性。
嚴格QC:表觀修飾性抗體檢測方案—多物種(H M R),多應用(DB WB IHC IF ChIP),出庫前全部經過內部嚴格QC檢測,極大程度滿足客戶需求。
抗體Kit:ABclonal表觀抗體全部自主研發,根據功能性研究對抗體進行組裝,提供系統性解決方案。
ChIP-Seq服務:ABclonal不僅能夠提供ChIP級抗體,同時能夠提供 ChIP 實驗、ChIP-qPCR 檢測、高通量測序文庫構建、生物信息學數據分析等全套服務。
新靶點合作開發:科學家鑑定出新的修飾位點或者發現新的修飾類型,ABclonal公司可以合作開發,鑑定成功,客戶可購買成品。
代表性抗體展示
TriMethyl-Histone
H3-K9 pAb
A2360 H3K9me3
Western blot
analysis of extracts of various cell lines, using TriMethyl-Histone H3-K9
antibody (A2360) at1:1000 dilution.
Dot-blot
analysis of all sorts of methylation peptides using TriMethyl-Histone H3-K9
antibody (A2360).
Immunohistochemistry
of paraffin-embedded Rat brain using TriMethyl-Histone H3-K9 antibody (A2360)
at dilution of 1:100 (40x lens).
at dilution of 1:100 (40x lens).
Chromatin
immunoprecipitation analysis extracts of 293T cells, using TriMethyl-Histone
H3-K9 antibody (A2360) and rabbit IgG.
P1 and P2 were located on EBAG9 gene. The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram was constructed by the ratios of the immunoprecipitated DNA to the input.
P1 and P2 were located on EBAG9 gene. The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram was constructed by the ratios of the immunoprecipitated DNA to the input.
TriMethyl-Histone
H3-K27 pAb
A2363 H3K27me3
Western blot
analysis of extracts of various cell lines, using TriMethyl-Histone H3-K27
antibody (A2363)
at 1:1000 dilution.
at 1:1000 dilution.
Dot-blot
analysis of all sorts of methylation peptides using TriMethyl-Histone H3-K27
antibody (A2363).
Immunofluorescence
analysis of HeLacells using TriMethyl-Histone H3-K27 antibody (Green) and BrdU
antibody (A1482) (Red).Blue: DAPIfor nuclear staining.
Chromatin
immunoprecipitation analysis extracts of 293T cells, using TriMethyl-Histone
H3-K27 antibody (A2363) and rabbit IgG. P1 and P2 were located on ANO2 gene.
The amount of immunoprecipitated DNA was checked by quantitative PCR. Histogram
was constructed by the ratios of the immunoprecipitated DNA to the input.
TriMethyl-Histone
H4-K20 pAb
A2372
Western blot
analysis of extracts of various cell lines, using TriMethyl-Histone H4-K20
antibody (A2372)
at 1:1000 dilution.
at 1:1000 dilution.
Dot-blot
analysis of all sorts of methylation peptides using TriMethyl-Histone H4-K20
antibody (A2372).
Immunohistochemistry
of paraffin-embedded rat testis using TriMethyl-Histone H4-K20 antibody (A2372)
at dilution of 1:200 (40x lens).